ゲノム研究の新たな道 ―微生物1細胞ゲノム解析用「AGM™試薬キット」の可能性
本記事の内容は、発行日現在の情報です。
製品名や組織名など最新情報と異なる場合がございますので、あらかじめご了承ください。
PDFダウンロード
PDFをダウンロードいただくには、会員登録が必要です
- 目次

生物の持つ遺伝情報、ゲノム。そのゲノム解析(1細胞ゲノム解析)のための新たな手法として東陽テクニカが発売する「AGM™(アガロースゲル・マイクロカプセル)試薬キット」の使用について、国立大学法人東京工業大学 生命理工学院 教授の本郷裕一氏よりお話を伺いました。AGMの作製技術は、国立研究開発法人理化学研究所によって研究開発されたもので、本郷氏も研究開発に参加されているほか、ユーザーとして「AGM™試薬キット」をご使用いただいています。
まずは本郷氏のバックグラウンドや近年のゲノム解析事情、1細胞ゲノム解析(シングルセルゲノミクス)とメタゲノム解析(メタゲノミクス)の違いなどに触れ、その後「AGM™試薬キット」の特徴やメリット、本郷氏が考えるAGMの今後の展望をご紹介します。
【インタビュアー】
小森研治
(株式会社東陽テクニカ ワン・テクノロジーズ・カンパニー インキュベーションユニット 課長)
虫取り網から次世代DNAシーケンサーへ
―「虫屋」本郷裕一氏

東京工業大学生命理工学院について教えてください。
東京工業大学生命理工学院は生物学・化学・物理学など多様なバックグラウンドを持つ教員が在籍し、60以上の研究室が存在する、生命科学分野としては国内最大級の学部・大学院です。分子生物学・タンパク質工学・遺伝子工学・微生物学などから、有機化学・生物物理学・バイオインフォマティクスなどにいたるまで、多岐にわたる生命科学分野の研究が行われています。

本郷教授の研究テーマや研究の道に進まれたきっかけについてお聞かせいただけますか。
私は先天的に虫が好きで、業界用語で「虫屋」と言います。子供の頃から虫を観察していて、気がついたら今でも同じことをやっている感じですね。道具が虫取り網からマイクロマニピュレーターや次世代DNAシーケンサーになっただけです。大学では、昆虫に共生する微生物を研究していましたが、そこから今は微生物を専門としています。
現在の主な研究対象はシロアリとその腸内微生物です。特に、数百種類もいる腸内微生物同士の共生関係に興味を持っています。シロアリは木材だけを食べて繁殖するユニークな動物ですが、木材の消化の大部分を腸内微生物が担っています。その微生物はシロアリの腸内のみに見られる系統群で、シロアリも腸内微生物同士も1億5,000万年以上にわたって共生関係を続けています。私はその複雑な共生メカニズムを最先端のツールを使って読み解こうとしています。
生命の設計図を読み解くゲノム解析
―昨今の状況・1細胞の微生物ゲノム解析
ゲノム解析の必要性とは?
遺伝子全体を「ゲノム」と呼びますが、それは生命の設計図でもあります。そのゲノムを解読することは、まさに生命の仕組みを読み解くことにほかなりません。知的好奇心を満たすためだけではなく、ヒトの病気のメカニズム解明や病原菌の感染メカニズムなどの理解をするためにもゲノム配列をベースに研究するのが当然の時代となっています。シロアリにおいては、腸内微生物が持つ木質分解酵素などのさまざまな有用遺伝子を探索するうえでゲノム解析が重要です。
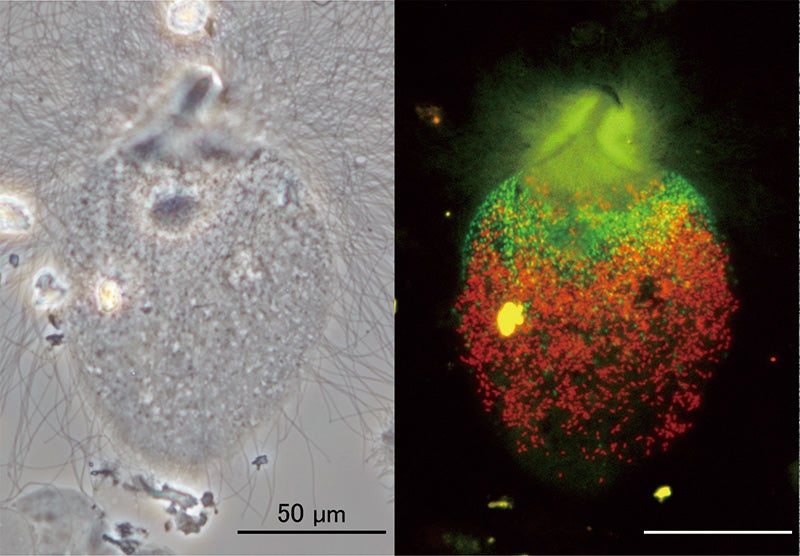
図1:シロアリ腸内原生生物と2種の細胞内共生細菌(本郷氏提供)
近年のゲノム解析技術の進歩について教えてください。
2000年代初期はゲノム配列を取得するコストが非常に大きいことから、ごく一部の研究者や研究機関しか実施できなかったため、解析対象もマウスなどのモデル生物から優先的に解読せざるを得ませんでした。
それが2000年代後半に次世代シーケンサー(従来型よりも高速かつ低コストで多くのDNAを解析できる装置)が登場し、比較的低コストでゲノム配列の取得が可能となりました。特に2010年代以後は、さらに低コストで高性能なシーケンサーが普及し、これによりモデル生物だけではなく多くの生物をゲノム解析の対象とできるようになりました。
動植物などの真核生物はゲノムサイズが大きいので現在でも解読は難しいのですが、バクテリアのゲノム解析であれば比較的容易にできるようになっています。
1細胞ゲノム解析とメタゲノム解析について教えてください。
次世代シーケンサーが登場したゲノミクスの時代にあっても難しかったことが、培養できない微生物の研究です。
培養できないあるいは難しい微生物は決して特殊なものではありません。むしろ、大腸菌や枯草菌、酵母など培養法が確立している微生物種の方が割合として全体の1%以下と非常に少ないです。
ゲノム配列の取得にはできればマイクログラム、少なくともナノグラム以上のDNA量が必要なので、純粋培養ができないと微生物のゲノム解析は物理的に不可能でした。そこでまず編み出された手法の一つが「メタゲノミクス」です。メタゲノミクスは環境中の微生物群集すべてのDNAを抽出して断片化し、それらの配列をできるだけ多く網羅的に取得することで群集全体としての機能を予測するものです。
しかし、この手法では、すべてのDNAを断片にして混ざったものを読むので群集全体のゲノムはわかっても、群集に含まれる個々の微生物種のゲノム配列を再構築することはできませんでした。
「シングルセルゲノミクス」とはその後に考案された手法です。メタゲノミクスとの違いは、はじめに物理的に細胞を分けてそこから出てくるDNAを見るため、どの生物種のゲノムか確実にわかるという点です。実際の手法としては、微生物細胞をマイクロマニピュレーターやセルソーターなどで物理的に単離し、Phi29というバクテリオファージ由来のDNAポリメラーゼを用いて「全ゲノム増幅」することでゲノム配列の取得を可能とします。
環境微生物を扱う分野では、この「シングルセルゲノミクス」と「メタゲノミクス」の2つの手法によって研究成果が爆発的に増え、研究者人口も大幅に増加しました。
1細胞ゲノム解析の新たな手法
―「AGM™試薬キット」について

ここからは、シングルセルゲノミクスをサポートする「AGM™試薬キット」を本郷氏が使用された感想や、今後の展望などについて紹介します。
「AGM™試薬キット」はゲノム解析にどう役立っていますか。従来までの方法との比較や、コスト面も含めてご教示ください。
図2:「AGM™試薬キット」
「AGM™試薬キット」の最大の長所は、大きな装置が必要なく、操作が簡単で職人芸的な技術は必要ない点です。まだ操作手順を改良する余地はありますが、練習すれば学生でもシングルセルゲノミクスを実行できます。
「AGM™試薬キット」を使えば、従来のように、購入に数千万円が必要な蛍光セルソーター(FACS:バクテリアを1細胞ずつ分け取る装置)や、作製に機械系の知識が必要なマイクロ流路デバイスなど、一般的な微生物研究者が入手困難なものを用いる必要がありません。
FACSは普段iPS細胞などの医学系研究で使用されることが多く、それを微生物研究のために使用させてもらうのは難しいです。そのため、微生物研究用として個々で購入する必要がありましたが、これを「AGM™試薬キット」のような使い捨てのキットで代替できる点は魅力です。
マイクロ流路デバイス作製については機械系の研究者と連携できれば可能ですが、そうでない場合は微生物研究者の多くが持ち得ない精密加工の専門的な知識・技術が必要となります。AGMを使用した場合でも特別な装置としてマイクロマニピュレーターだけは必要となりますが、これは普通の研究者であっても購入が可能な範囲と言えます。
全ゲノム増幅試薬や配列解析などゲノム解析に関係するコストは従来通り必要なものの、FACSのような特別な装置は不要、また、「AGM™試薬キット」を構成する主な試薬は生物系の実験において一般的な部類のものであるため、解析に必要なゲノムを取得するコストは通常の生物学・化学実験にかかるコストと同等であることはメリットです。
また、私の研究テーマであるシロアリの腸内細菌を研究するうえでは、AGMにより共生しているバクテリア同士をペアの状態で単離できる点がこれからの研究で役立つと考えています。
「AGM™試薬キット」を使って1細胞でゲノム解析を行う理由や、1細胞ゲノム解析とメタゲノム解析がどのように相補するのかについて、もう少し踏み込んでお聞かせください。

結論をいうと、メタゲノミクスは特別な技術が不要なため広く普及していますが、欠点もあるためシングルセルゲノミクスと両方行うほうがよいでしょう。
近年、メタゲノミクスはコンピューター上で配列断片を細菌種ごとに仕分けする「ビニング」と呼ばれる手法が発達し、無料で入手できるプログラムを使って誰でも手軽に個々の細菌種のゲノムをある程度再構築できるようになっています。しかし、そうして仕分けされた配列断片はあくまでもプログラムによって情報学的に分類されたもので、本当に正しいものかどうかはわかりません。丁寧に配列を調べてみると、誤って分類されてしまっている断片も少なくなく、また分類マーカーとして長年使用されてきたリボソームRNA遺伝子の配列を正しく仕分けるのが難しいという、研究者からすると致命的な欠点もあります。
また、細菌細胞はバクテリオファージに感染していたり、染色体の遺伝子とは異なるプラスミドというDNAを持っていたりすることが多いのですが、それらの可動因子もメタゲノミクスでは仕分けが困難です。そうした欠点を補うためにもシングルセルゲノミクスが役に立ちます。
シングルセルゲノミクスでは物理的に細胞を単離しますので、より確実に種ごとのゲノム配列再構築が可能となり、リボソーム遺伝子や可動因子も解析することができます。メタゲノミクスでは解析しにくい群集の中でマイナーな微生物種のゲノム配列を再構築できる可能性も高くなるでしょう。また、メタゲノミクスと違って、シングルセルゲノミクスはゲノムサイズが大きい真核生物にも適用できます。
実際、哺乳類のシングルセルゲノミクスは盛んに行われています。例えばがん細胞は一つ一つのゲノムの構成が異なるのですが、その細胞ごとの変異を解析することも可能となっています。
環境微生物の分野においては、染色体外遺伝子(プラスミド)も捕捉できるようになることに大きな意義があり、生態学的に重要なことや抗生物質耐性遺伝子に関係することも解析できるようになります。
AGMの今後について期待されることはどのような点でしょうか。

図3:AGMイメージ
現在も私の研究室でAGM作製を行っていますが、必要試薬量の最小化やAGMのサイズの均一化など、まだ改良の余地があると思います。具体的には50mLチューブを使用している手順を1.5mLチューブで済むようにする、などですね。
また、標的とする細菌種のゲノム増幅産物を含むAGMを識別できるようになることにも期待したいです。標的を含んだAGMの見分けがつかないと、DNA増幅が見られたAGMを片端からゲノム解析することになってしまいます。
より幅広いユーザーに使用してもらうためという観点では、コストは増えますが個々の増幅産物をバーコーディングするなどしてマニピュレーションせずに網羅的な配列解析ができるようにすることや、FACSを利用することでスループットを上げるための組み合わせも応用例として提示できると良いかもしれません。
細胞をAGMに包埋してからFACSを利用するのと、細胞をそのままでFACSを利用するのとでは、何か違いがあるのでしょうか。
そこには大きな違いがあります。従来のように細胞をそのままでFACSを利用すると、マイクロリットルオーダーの(広い)空間でゲノム増幅させることになり、均質な増幅が得られないということが課題となっていました。一方、ピコリットルオーダーの(狭い)空間でゲノム増幅させると、ゲノム全体にわたって均質に増幅することが知られていて、AGMに包埋することで、それを実現できるのです。
AGMで細胞を単離した後、マイクロマニピュレーターで一つ一つをピックアップした場合は数十個単位の解析になりますが、FACSを使用した場合だと自動的に数百や数千単位まで解析ができますので、スループットを上げることにつながります。
また、AGMで処理せずにFACSでソートした場合は増幅されやすいDNAと増幅されにくいDNAが現れるのですが、AGMで微生物を単離した後にFACSでソートできれば、増幅されたDNAが均一になり、より正しい解析が可能となることにもつながるでしょう。
一研究者として大切にしていること

先生が研究者として大事にされていることを教えていただけますか。
研究とはアートだと思っています。どういう研究テーマを選ぶのか、どこまで掘り下げるのか、どれだけ証拠を揃えるのか、これは個々の研究者次第であり、それが研究を職業とする人間の自己表現です。
一方で、我々はプロの研究者ですから、現実的でもなければなりません。特に大学教員は学生に論文を出させるという使命があるので、なかなか理想通りにはいきません。それでも、私自身を納得させることができない、疑問が残る結論をあたかも真実であるかのように言ってしまう論文は出させないですね。
これからの若い研究者に向けて何かメッセージをいただけませんか。
月並みですけど幅広い分野の研究者と積極的に交流してほしいですね。
私自身のバックグラウンドは動物学(=虫)と分子生物学だったのですが、微生物学、生態学、それに情報科学や機械工学の専門家たちと交流する中で、自然と微生物生態学やゲノム微生物学そして1細胞ゲノミクスを専門とするようになりました。さまざまな研究者と交流することで融合するものがありますので、オープンにいろんな研究者と交流しましょう。
最後に、日本のゲノム解析とAGMの今後についてお聞かせいただけますか。
ゲノミクスの分野だけではないですが、日本人は良くも悪くもこだわりが強く、職人芸的な細かなことが好きなので、大まかな側面がある「メタ」解析は苦手です。それにゲノム配列取得には費用が必要ですが、日本の普通の研究者には潤沢な資金がありません。そのため、2000年代後半からの世界的なメタゲノミクスブームから完全に取り残されてしまい、それを今でも引きずっています。
シングルセルゲノミクスについては元々世界的に実施できる研究室が限定されていたので、日本だけが特に遅れて、ということはありません。ゲノミクスをより一般的にするためにも、AGMのような低コストの手法の開発が一層進むことを期待します。


